This is a translation in Danish. You can also read the original English version.
COVID-19 HGI resultater, 2. juli 2, 2020
September 25, 2020
Skrevet på engelsk af Brooke Wolford og Kumar Veerapen og oversat af Lasse Folkersen og Ida Moltke - på vegne af COVID-19 HGI
Disclaimers: For det første - dette er igangværende forskning. Selvom vi allerede opdager ting, har vi også brug for flere prøver for at få en robust forståelse af hvordan den menneskelig genetik bidrager til COVID-19 infektioner. Jo flere prøver vi tilføjer til vores studie, jo mere sikre er vi på at disse mønstre faktisk eksisterer og er repræsentative på tværs af forskellige grupper af patienter. For det andet - vi er ikke i stand til at fortælle dig noget om risikoen for at få alvorlig COVID-19 blot på baggrund af din genetik. Brugerne af vores resultater bør ikke bruge vores fund til at diagnosticere COVID-19 patienter ud fra deres genotype, og bør altid tale med en sundhedsfaglig person for at rådgive i medicinske spørgsmål. Til sidst - hvis der er nogle ord vi bruger som ikke er forståelige, så venligst send en email til hgi-faq@icda.bio - vi vil med glæde opdatere informationen her så den fremstår mere klart. I de kommende uger vil yderligere information frigives, som forklarer disse koncepter. I mellemtiden, så brug gerne f.eks. dette website om basal genetik.COVID-19 pandemien har påvirket hverdagen for mennesker i hele verden. Forskere overalt arbejder derfor hårdt for bedre at forstå virussen og den sygdom den forårsager. Vi repræsenterer en sådan forskningsgruppe - COVID-19 Host Genetics Initiative (HGI) - et internationalt team af genetikere, som er fokuserede på at identificere de menneskelige genvariationer, som kan påvirke, hvordan vi reagerer på SARS-CoV-2 infektion og den deraf følgende sygdom, COVID-19. I samarbejde vil vi prøve at forstå hvad det er i et menneskes DNA som kan påvirke hvorvidt nogen udvikler COVID-19, og hvis de gør, hvor syge de så bliver.
COVID-19 HGI studiets design
I vores forskningsprojekt sammenligner vi genetiske variationer mellem patienter, dvs. folk som er blevet indlagt og har en positiv COVID-19 test, med raske kontroller, altså folk fra den bredere befolkning som ikke har en positiv COVID-19 test. En sådan sammenligning kaldes for et Genome Wide Association Study eller GWAS. Hvad det præcis er er forklaret bedre andetsteds, f.eks. denne her eller her. Per juli 2020 har vi samlet resultater fra otte forskellige studier, som i alt inkluderer 3199 patienter og 897488 raske kontroller.
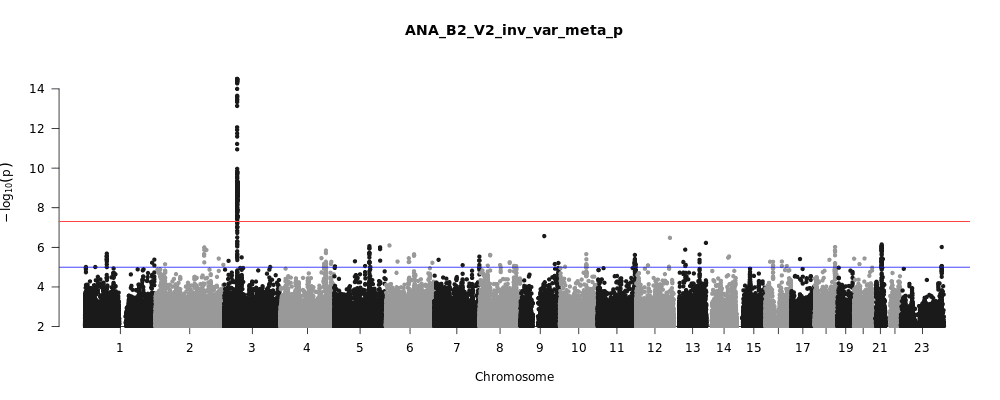
COVID-19 HGI sammenkæder genetiske varianter med alvorlighed af COVID-19
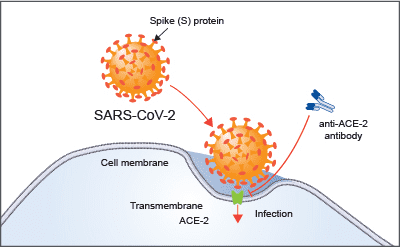
Figur 1, herover, opsummerer de seneste resultater fra COVID-19 HGI studierne. Denne figur-type kaldes et Manhattan plot. Hvad det betyder er nærmere i forklaret i fodnoten, men dybest set bruges et Manhattan plot til at visualisere statistiske sammenhænge, også kaldet associationer, mellem genetiske varianter og de træk som studeres (her f.eks. COVID-19). Vi observerede en genetisk region på kromosom 3 med genetiske varianter, som var statistisk signifikant associeret med COVID-19 (som det ses ved prikkerne over det sted hvor kromosom 3 er angivet på X-aksen). Det hænder at en region inkluderer flere gener som findes tæt på hinanden i det pågældende område af genomet, og det vil kræve mere forskning at finde ud af præcis hvilket gen som er ansvarlig for den øgede alvorlighed af COVID-19 i de mennesker som bærer risiko-varianten. Regionen på kromosom 3 er af den type som overlapper flere gener, præcis hvilke er angivet i figur 2. Det er uklart hvilke af disse gener i denne region som faktisk er ansvarlige. Vi har dog nogle interessante ledetråde! I regionen findes flere gener som beskriver de såkaldte kemokiner, f.eks. generne CXCR6 og CCR1. Kemokiner kontroller immun-cellers bevægelser rundt i kroppen og er strengt nødvendige for at det innate immunsystem kan fungere. Genet SLC5A20, som også findes i dette område, beskriver et protein som man ved binder til ACE-2. ACE-2 er det protein, som fungerer som en slags dør for SARS-CoV-2 virussen når den tiltvinger sig adgang til vores celler (figur 3). Det betyder at det muligvis også kan være en genetisk variant ved SLC6A20 genet, som gør det nemmere for virus at bryde ind i vores celler. Disse er blot de første eksempler fra vores forskningsprojekt.
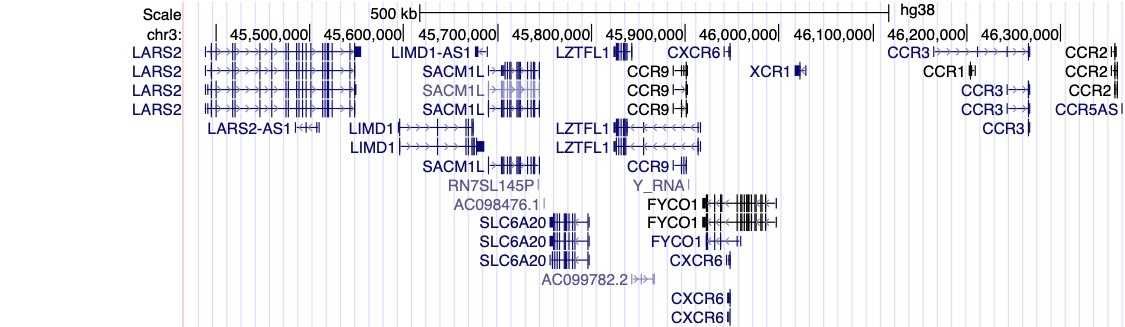
Sammenligning af vores resultater med resultater fra andre studier.
Som du måske har hørt i nyhederne, har det været rapporteret at blodtype har haft nogen korrelation med COVID-19, således at mennesker med blodtype A skulle have en højere sygdomsrisiko og mennesker med blodtype O skulle have en lavere sygdomsrisiko. Et nyligt studie fra New England Journal of Medicine (NEJM) beskrev hvordan de havde fundet dette i 1980 deltagere fra Italien og Spanien, og senere replikeret resultaterne hos det amerikanske DNA-firma 23andme. I disse studier så det ud til at ABO grupperne på kromosom 9 har signifikant betydning for udviklingen af COVID-19. I disse studier er der dog blevet brugt bloddonorer som kontrolgrupper, og bloddonorer har ofte blodtype O. De er derfor ikke et særligt godt sammenligningsgrundlag. I vores studie, i figur 1, Manhattan plottet, kan man da heller ikke se nogen sammenhæng med COVID-19 i det område af kromosom 9, og det selvom data fra det oprindelige NEJM studie faktisk også er med i analysen. Vi har derfor konkluderet at der kræves flere prøver for at kunne sige om denne ABO region af genomet faktisk hænger sammen med COVID-19.
Anerkendelse af vores forskningsprojekts begrænsninger
Intet studiedesign er perfekt og vi vil gerne gøre opmærksom på et par begrænsninger ved vores forskningsprojekt. For det første er de resultater, der er beskrevet ovenfor, foreløbige og baseret på det data vi havde tilgængeligt i juli 2020. Selvom vi har nok data til at lave nogle indledende observationer, vil fremtidige gentagelser af vores analyser med flere studiedeltagere hjælpe os til at få større tiltro til vores konklusioner. Desværre vil flere studiedeltagere betyde at flere mennesker er blevet smittet med COVID-19, men det vil også forbedre vores evner til at finde sammenhænge mellem genetik og reaktion på COVID-19.
For det andet kan definitionen af alvorlighed af COVID-19 variere fra et studie til et andet. Desuden har vi antaget at kontrollerne ikke har COVID-19, men vi ved at der er mange asymptomatiske sygdomsbærere i samfundet, så nogle af disse “raske kontroller” har muligvis COVID-19. Disse begrænsninger kan overkommes ved at øge antallet af både patienter og kontroller der deltager i studierne: jo flere studiedeltagere vi analyserer data fra, jo lavere er risikoen for at observere falske positiver på grund af begrænsningerne i vores studiedesign. Og så snart et positivt associationssignal er identificeret, så kan vi fokusere på et mindre studie med mere specifikke definitioner af patient- og kontrol-grupperne for at validere signalet. I sidste ende kræver det yderligere forskning at bruge vores genetiske fund til at opnå indsigt i sygdomsmekanismerne.
Vores næste skridt
For at gøre noget ved den nuværende begrænsning i deltagerantal bliver vi ved med at indsamle og analysere yderligere data fra de studier der er med i forskningsprojektet. Den næste runde analyser vil blive udført i slutningen af september og resultaterne vil blive gjort tilgængelige i starten af oktober 2020. Vi håber på at opnå ny viden fra de kommende resultater, da vi regner med at have data fra op til 50% flere studiedeltagere. Vi regner også med at indsamle mere detaljeret information om COVID-19 patienternes symptomer. Kom her tilbage og check hvad vi har lært i oktober!
Ved at bruge vores foreløbige resultater kan detektivarbejdet begynde. Vores forskningsgruppe og andre forskere kan udføre yderligere studier for bedre at forstå de biologiske processer der er påvirket af de gener vi har fundet og hvordan det muligvis er relevant for reaktionen på COVID-19. Hvis du vil have flere detaljer om disse opfølgningsstudier, så følg dette link. Et af disse studier vil undersøge hvilken association der er mellem de genetiske varianter vi har fundet og sygdomsforløbet særligt hos de hospitalspatienter, der er hårdest ramt. Vi glæder os til at få en bedre forståelse af vores genetiske fund og håber på at det kan føre til bedre klinisk håndtering af COVID-19 patienter eller sygdomsbehandlinger.
Yderligere ressourcer
Hvis du vil læse mere om COVID-19 Host Genetics Initiative, kan du finde mediedækning i de daglige aviser:
Tak
Tak til Rachel Liao, Caitlin Cooney, CGC, Karen Zusi, Andrea Ganna, Alina Chan, Sophie Limou, Shea Andrews og Jamal Nasir for gode kommentarer og forslag til ændringer.
Fodnote
Et Manhattan plot (navnet skyldes at toppene tit får plottet til at ligne New Yorks skyline) er en ofte brugt måde at visualisere GWAS resultater på. Den vandrette linje, eller X-aksen (“Chromosome”), viser positionen af genetiske varianter langs de 23 kromosomer (mennesker har 22 kromosompar plus en kombination af X og Y sexkromosomer). Den lodrette linje, eller Y-aksen, viser et mål for statistisk signifikans, som kaldes p-værdi, transformeret til negativ logaritmisk skala. Hvert punkt i plottet illustrerer den statistiske signifikans (p-værdien) for at der er en association mellem en genetisk variant på en given position i genomet (ofte kaldet en SNP, udtalt “snip”) og et givet træk målt for hver deltager. Jo højere et punkt ligger på den lodrette akse, jo mere sandsynligt er det at denne SNP er associeret med det relevante træk (f.eks. alvorlighed af COVID-19). Den metode vi har brugt er konservativ: hvor mange studier kræver en p-værdi på under 0.05 for at anse en association for at være statistisk signifikant, kræver vi istedet en p-værdi på under 0.00000005 (vist med den røde vandrette linje) for at forbedre vores tiltro til de signaler vi finder. Det vil sige at hvis punkterne er højere end den røde linje, anser vi den genetiske association for at være “statistisk signifikant” og dermed værd at designe eksperimenter, der yderligere kan validere og som kan hjælpe os med at forstå SNPens biologiske relevans.
