This is a translation in Swedish. You can also read the original English version.
COVID-19 HGI resultat, daterade 2.7.2020
September 25, 2020
Brooke Wolford och Kumar Veerapen, på uppdrag av COVID-19 HGI
Ansvarsfriskrivning: För det första, vänligen observera att denna forskning är pågående. Samtidigt som vi redan gör upptäckter, behöver vi mera prov för att få en robust förståelse för den genetiska medverkan i COVID-19 resultaten. Ju mera prov vi lägger till i vår studie, destu mer säkra kan vi vara på att de mönster vi observerar existerar och är representativa i olika patientgrupper. För det andra, vi kan inte förutse vilken sannolikheten är för en allvarlig COVID-19 beroende på din genetik. Användare av våra resultat skall därför inte använda våra upptäckter för att diagnostisera patienter utgående från deras genotyp och skall alltid diskutera med medicinska experter för vägledning av medicinsk vård. Slutligen, om vokabuläret här är obekant, vänligen skicka epost till hgi-faq@icda.bio– vi hjälper gärna med att klargöra informationen i den bifogade texten. Mer information gällande terminologin kommer att bli tillgänglig inom kommande veckor. Under tiden, läs this källa för sammanfattning av grundläggande genetik.COVID-19 pandemin har globalt påverkat påverkat människors vardag. Forskare världen över har jobbat hårt för att bättre förstå viruset och sjukdomen. Vi representerar en dylik grupp – COVID-19 Host Genetics Initiative (HGI) – ett internationellt team av genetiker, som fokuserar sig på att identifiera mänskliga genetiska variationer, vilka i sin tur påverkar responsen av SARS-CoV-2-infektionen samt den påföljande sjukdomen, COVID-19. Vårt gemensamma intresse är att utreda vilka avsnitt i människans DNA, som kan påverka att en person utvecklar COVID-19 och ifall de gör, hur sjuka de blir.
Forskningsplan för COVID-19 HGI
I vår studie jämför vi den genetiska variationen mellan patienter, dvs personer som har blivit inlagda på sjukhus och testat positivt för SARS-CoV-2, och kontroller, som är personer från den allmänna populationen, vilka inte testat positivt för COVID-19. Den här jämförelsen kallas för en Genome Wide Association Study (GWAS). Titta på denna video eller denna informationsgrafik för en illustrerad beskrivning av GWAS! Daterat juli 2020 har vi ett kombinerat resultat baserat på åtta olika studier gällande totalt 3,199 patienter och 897,488 kontroller.
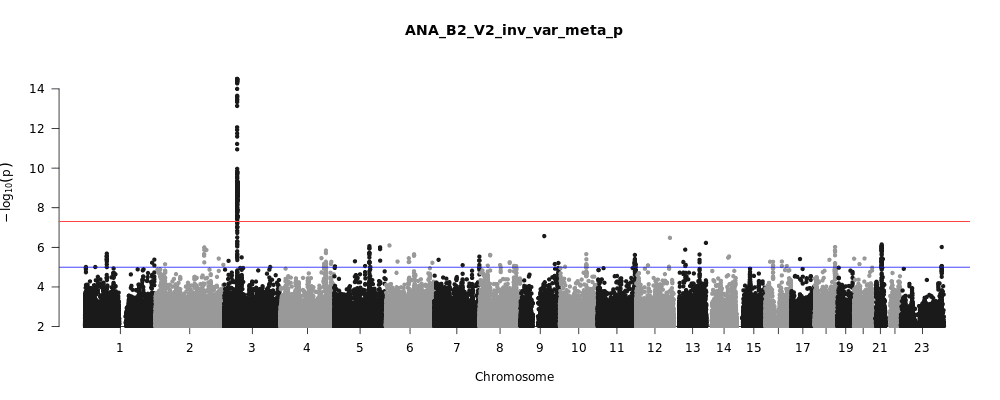
COVID-19 HGI upptäcker genetisk variation associerad med svårighetsgrad av COVID-19
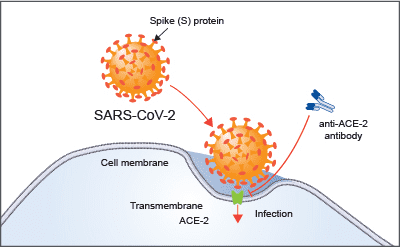
Figur 1 ovan visar en visuell sammanfattning av de senaste resultaten från COVD-19 HGI. Detta kallas för en ”Manhattan plot”, vänligen läs fotnoten för en omfattande beskrivning av visualiseringen. Kortfattat, en ”Manhattan plot” används för att visa association mellan en egenskap (tex COVID-19) och genetiska varianter över hela genomet. Vi observerar ett statistiskt signifikant område på kromosom 3 (observera den punktstreckade, vertikala linjen ovanför kromosom 3, som är indikerad på den horisontala X-axeln). Ibland inkluderar en region ett flertal närliggande gener. Det kräver fortsatt forskning för att begränsa den statistiskt signifikanta regionen till en specifik gen, som är involverad i svårighetsgraden av COVID-19. Den identifierade regionen på kromosom 3 överlappar med flera gener( se en lista på alla gennamn i Figur 2). Det är inte klarlagt vilken specifik gen inom denna begränsade region, som är associerad med svårighetsgraden av COVID-19. Hur som helst så har vi några intressanta ledtrådar! Det finns ett flertal kemokin-relaterade gener inom detta område, så som CXCR6 och CCR1. Kemokiner kontrollerar immuncellernas rörelse och är kritiska för att det medfödda immunförsvaret skall fungera ordentligt. Genen SLC6A20 finns också i denna region, och den tillverkar ett protein som är känt för att binda till ACE2. Proteinet ACE2 i sin tur fungerar som en dörr, vilket SARS-CoV-2 använder som ingång till våra celler (Figur 3). Detta betyder att det är möjligt att genetisk variation hos SLC6A20 påverkar viralt inträde! De här resultaten från våra upptäckter av genetisk association är endast det första steget i forskningsprocessen.
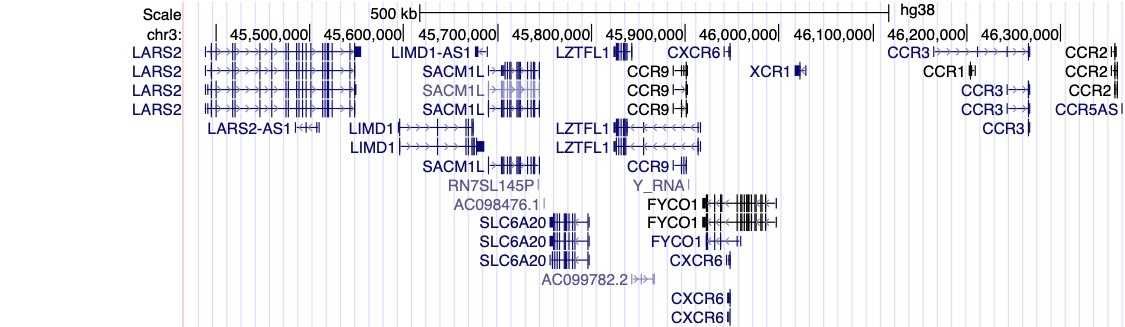
Jämförelse av våra resultat mot resultat från andra studier
Du har kanske hört via nyheterna att blodgrupper verkar vara associerade med COVID-19, så att typ A korrelerar med en högre risk och typ O fungerar som skyddande. En nyligen utkommen forskningsartikel i tidskriften New England Journal of Medicine (NEJM) beskriver en genetisk associationsanalys för allvarlig COVID-19 (tex intagen på sjukhus med andningssvikt) innefattande 1,980 personer från Italien och Spanien (och även replikerad av 23andMe). I denna studie verkar ABO-blodgrupperna på kromosom 9 vara signifikant associerade med COVID-19. Hur som helst, denna studie använde bloddonatorer som kontrollgrupp för analysen, och dessa tenderar att ha flera typ O-individer, vilket eventuellt inte är en idealisk jämförelse för personer, som har smittats med COVID-19. Detta bekräftas i vårt data: i ”Manhattan plotten” i Figur 1 ser vi inget signifikant resultat för kromosom 9 ( dvs inga punkter som stiger ovanom den röda linjen). Detta betyder att COVID-19 HGI analysen, som inkluderar data från NEJM, inte bekräftar associationerna med ABO-blodgruppsgenerna i detta skede. Vi behöver ett större antal prov för att klargöra om denna region är associerad med COVID-19.
Tillkännagivande av begränsningar i vår studie
Ingen forskningsplan är perfekt och vi vill klargöra några begränsningar i vår forskning. För det första, våra ovan beskrivna resultat är preliminära och baserade på data från juli 2020. Vi har tillräckligt med data för att göra begynnande observationer, men större antal prover i framtida upprepningar kommer att göra oss mera säkra på våra konklusioner. En större provstorlek innebär tyvärr att flera personer måste bli infekterade av SARS-CoV-viruset, men det innebär samtidigt förbättrade möjligheter att hitta mönster mellan värdgenomet och sjukdomsutgången.
För det andra, definitionen av sjukdomens svårighetsgrad kan variera från en individuell studie till en annan. Därtill antar man att kontrollerna inte har haft COVID-19, men vi vet att det finns många asymptomatiska individer, vilket innebär att en del av dessa ”kontroller” kan ha smittats av COVID-19. Hur som helst, dessa begränsningar kan övervinnas genom att öka antalet patienter och kontroller som analyseras: ju flera prover som analyseras, destu lägre är risken för att observera falskt positiva signaler i studien. När sedan en positiv signal identifieras, kan vi fokusera på mindre studier med mer specifika begränsningar för patient- och kontrollgrupper för att validera våra resultat. Sammanfattningsvis, det krävs mera forskning för att klarlägga de sjukdomsmekanismer som våra genetiska resultat uppvisar.
Våra nästa steg
För att utöka provstorleken tar vi kontinuerligt emot information från medverkande studier. Nästa analys kommer att göras i slutet av september och resultaten kommer tidigast att ges ut i början av oktober. Vi hoppas på att erhålla mera information gällande promises att ha en provstorlek på upp till 50% av det vi har nu. Vi ser också fram emot att samla in mer omfattande och detaljrik data om symptom, som COVID-19-patienterna har. Kolla pånytt här för att läsa om vad vi har lärt oss i oktober 2020!
Genom att använda våra preliminära resultat börjar detektivarbetet. Vårt konsortium och andra forskare kan genomföra flera studier för att bättre förstå de biologiska processer som påverkas av dessa gener och hur detta kan vara relevant för utgången av COVID-19. Om du vill ha mera uppgifter om påföljande studier, fortsätt till denna länk. En sådan studie kommer att undersöka hur den genetiska variationen är associerad med utgången speciellt hos svårast insjuknade patienterna, som behövt sjukhushusvård. Vi är ivriga att förstå våra genetiska resultat och hur de i framtiden kan leda till bättre klinisk behandling av COVID-19-patienter eller en bättre sjukdomsbehandling.
Övriga källor
För att läsa mera om COVID-19 Host Genetics Initiative, kolla mediebevakningen i följande tidskrifter
Tillkännagivanden
Tack till Rachel Liao, Caitlin Cooney, CGC, Karen Zusi, Andrea Ganna, Alina Chan, Sophie Limou, Shea Andrews och Jamal Nasir för eftertänksam respons och revision.
Fotnot
En Manhattan plot (namngett efter att topparna påminner om en silhuett av New York City-himlen) är en allmän visualisering av GWAS-resultat. Den horisontala linjen eller X-axeln (”Chromosome”) visar de genetiska varianternas positioner längs med de 23 kromosomerna (människan har 22 par av kromosomer samt en kombination av könskromosomerna X och Y). Den vertikala linjen eller Y-axeln visar ett mått på statistisk signifikans, kallat för p-värde, omformat i en negativ logaritmisk skala. Varje punkt på figuren visar den statistiska signifikansen (p-värdet) för associationen mellan en genetisk variant på en given kromosomposition (kallas för SNP, ”snip”) och sjukdomsbilden hos en undersökt person. Ju högre punkten befinner sig på den vertikala skalan, destu högre är sannolikheten för att den SNP:en är associerad med utfallet av intresse ( tex. COVID-19 svårighetsgrad). Vår metodologi är varsam: många studier kräver ett p-värde på mindre än 0.05 för att fyndet skall anses som signifikant, vi kräver ett p-värde på 0.00000005 (indikerat av den röda linjen) för att öka säkerheten i våra fynd. Om punkten är högre än den röda linjen, anser vi att den genetiska associationen är ”statistiskt signifikant” och vi kan därmed planera experiment för att vidare utreda och förstå SNP:ens biologiska relevans.
